大量ゲノム時代の比較ゲノム解析ツール「DiGAlign」の公開 ―「微生物ダークマター」の解明のためのwebサーバー―
京都大学化学研究所 山田航平 学部生(当時)、岡嵜友輔 助教、緒方博之 教授、国立研究開発法人海洋研究開発機構(JAMSTEC) 海洋機能利用分野生命理工学センター 西村陽介 特任研究員らの研究グループは、高機能なシンテニーマップ表示機能により、迅速な比較ゲノム解析ができるウェブツールDiGAlign(https://www.genome.jp/digalign/)を公開しました。
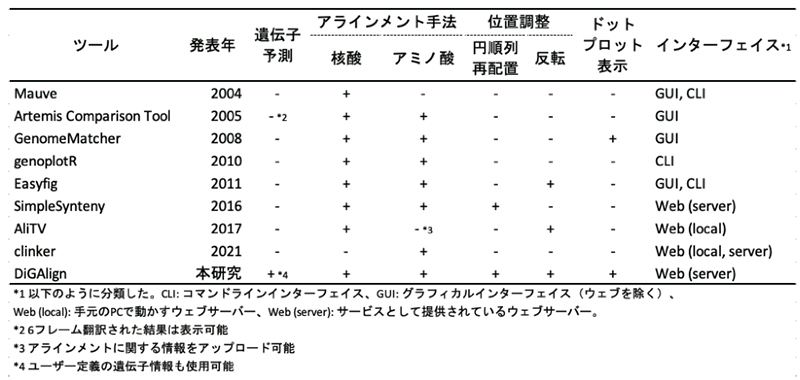
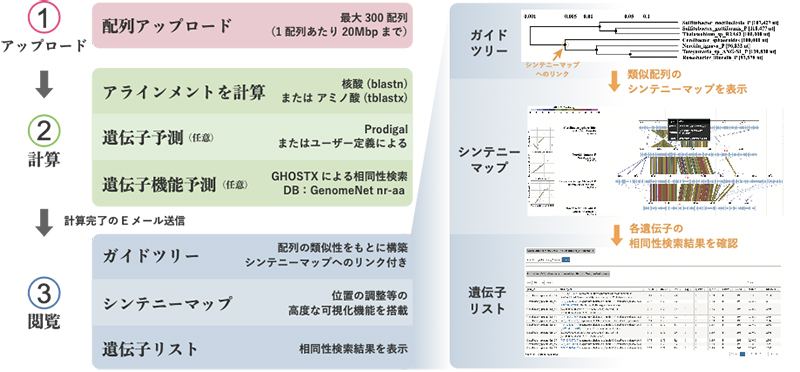
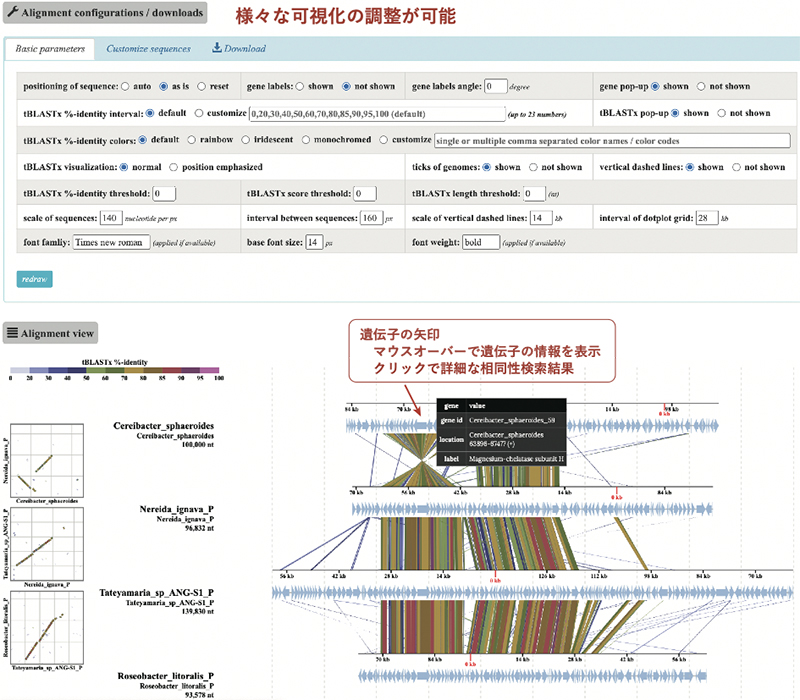
DiGAlignは、一度に最大300ゲノムという大規模な比較ゲノム解析が可能であり、これまでのシンテニーマップ作成ツールでは不十分だった様々な機能を備えています(図1)。特に、独自の機能として、類似性をもとに構築されるゲノム系統樹である「ガイドツリー」を利用して、良く似ているゲノムのシンテニーマップを簡単に表示できます(図2)。また、DiGAlignでは遺伝子の同定とその機能予測が行われ、事前の遺伝子解析を省略した迅速な比較ゲノム解析が可能です。さらに、シンテニーマップは様々な可視化パラメーターを備え、表示する配列の変更や、位置の調整、順番の入れ替えも可能です(図3)。
DiGAlignはこのような機能によって、仮説や事前知識なしにゲノムデータに解釈を立てることを可能とし、大量のゲノム情報を入手可能な現代におけるゲノム解析のスピードを加速します。微生物の大部分を占める未培養微生物は、「微生物ダークマター」と呼ばれその実態が解明されていません。DiGAlignはゲノム情報にもとに、個々の微生物が持つ物質生産・エネルギー変換の潜在能力や、微生物の進化や生態学的な役割の解明を推進します。
微生物のほとんどは未培養であり、未培養微生物はその実態が謎に包まれていることから、「微生物ダークマター」と呼ばれています。次世代シーケンサーの技術的な発展により、メタゲノム解析によってそのような微生物のゲノムを大量かつ安価に解読できる時代となりました。しかし、解読されたゲノム情報の活用には、多くの課題が残されています。
ゲノム情報から個々の微生物の活動や、その生態、進化を理解するためには、ゲノムを比較してその類似点や相違点を明らかにする「比較ゲノム解析」が有効です。しかし、比較ゲノム解析には専門的な解析技術が求められる上に、長大な解析時間を要することがあります。
比較ゲノム解析における可視化手法として、ゲノム間で類似する領域を図解する「シンテニーマップ」が一般的に利用されており、その作成ツールは数多く開発されてきました(図1)。しかし、大量のゲノムが入手可能な現在では比較すべきゲノムの組み合わせを最適化する必要があります。これまでのツールでは組み合わせを最適化するために繰り返し計算をかけることが求められ、効率の良いゲノム比較は困難でした。
本研究では、比較ゲノム解析を行うウェブツール「DiGAlign」を開発しました。DiGAlignでは、比較対象とするゲノム配列のアップロードにより、一度に最大300ゲノムという大規模な比較ゲノム解析が可能です。DiGAlignは、これまでのシンテニーマップ作成ツールにはない様々な機能を備えています。特に、独自の機能として、類似性をもとに構築されるゲノム系統樹である「ガイドツリー」が作成されます(図4)。ガイドツリー上の分岐点に配置されているリンクによって、その分岐点下に含まれる、類似性の高い(つまり、比較対象として適している)ゲノムのシンテニーマップを表示できます。また、シンテニーマップは様々な可視化パラメーターを備え、表示する配列の変更や、順番を入れ替えることも可能です(図3)。

発表者らは、ウイルスゲノムの比較と分類のためのウェブツールViPTreeを2017年に発表しており、現在最も良く利用されるウイルスゲノム分類ツールの一つとなっています。DiGAlignの開発ではViPTreeの技術基盤を活用し、インタラクティブな可視化のためにJavaScriptライブラリのd3.jsを使用しました。DiGAlignはウェブブラウザ上でデザインや配色の調整もできる可視化システムとなっており、論文の図としてそのまま使用可能なシンテニーマップを作成し、ベクター画像であるSVG形式でダウンロード可能です。
「微生物ダークマター」と呼ばれる未培養微生物は、様々な物質・エネルギー変換のポテンシャルを有しています。ゲノム情報から個々の微生物の活動を理解することは、新しいバイオ技術の開発や、地球環境の形成・維持の過程の解明につながります。DiGAlignは、高機能かつ迅速な比較ゲノム解析を可能とすることで、個々の微生物が持つ物質・エネルギー変換のポテンシャルの理解や、微生物の進化、生態学的な役割についての理解を促進します。筆者らは海洋微生物5万ゲノムのカタログを公開するなど、大規模なゲノムリソースの整備に取り組んでいます。このように大量のゲノム情報を入手可能な現代において、DiGAlignは事前の仮説や知識がなくてもゲノム情報に解釈を与えることを可能にし、地球上の微生物圏の理解に貢献します。
本研究は、日本学術振興会科研費(JP22K15089、JP22H05714、JP22H00384、JP22K15182)、科学技術振興機構ACT-X(JPMJAX21BK)、CREST(JPMJCR23B1)、京都大学化学研究所 国際共同利用・共同研究拠点(課題番号:2023-38) の支援により実施されました。また、本ウェブツールは京都大学化学研究所スーパーコンピュータシステムを利用しています。
●用語解説●
比較ゲノム解析:複数のゲノムを比較してその類似点や相違点を明らかにする解析。例えば、共通の遺伝子または独自の遺伝子を同定することで、それぞれのゲノムの代謝の特色や、進化の過程を考察できる。
微生物ダークマター:地球上に存在するほとんどの微生物は培養されておらず、その実態が謎に包まれていることを意味する。微生物は全部で1兆種に達し、その99.999%が未発見であると推定されている。最近では、未培養のまま微生物のゲノムを解読するメタゲノム解析によって、微生物ダークマターのゲノム解読が進んでいる。
 京都大学 化学研究所
京都大学 化学研究所 国際共同利用・共同研究拠点
国際共同利用・共同研究拠点