加藤 有己 特定研究員、阿久津 達也 教授ら「超高速なRNA間相互作用予測法を開発」(2010年9月「Bioinformatics」で発表)
平成22年9月 トピックス
加藤有己 特定研究員、阿久津達也 教授ら
(バイオインフォマティクスセンター 生物情報ネットワーク研究領域)

加藤有己特定研究員(左)と阿久津達也教授(右)
(2010年9月「Bioinformatics」で発表)
加藤有己特定研究員(京都大学化学研究所)、佐藤健吾特任講師(東京大学)を中心に、浜田道昭博士(みずほ情報総研、産業技術総合研究所)、渡邊芳英教授(同志社大学)、浅井潔教授(東京大学、産業技術総合研究所)、阿久津達也教授(京都大学化学研究所)からなる研究グループは、期待精度最大化原理と整数計画法の融合により、計算機に基づく手法としては従来の常識を打ち破る非常に高速なRNA間相互作用予測法の開発に成功しました。
RNA(リボ核酸)は私たち人間を含めた生物の細胞の中に存在している鎖状の分子です。一昔前までは、RNAはDNAの遺伝子からタンパク質を作るための情報のコピーとも言うべきメッセンジャーRNA(mRNA)などの、受動的な役割のみを持つとの認識が主流でした。
ところが近年、ノンコーディングRNA(ncRNA)と呼ばれるRNAのクラスの一部が遺伝子発現の転写後調節機構を持つことが指摘され、生物の仕組みを知るためには無視できない要素の1つとしてクローズアップされています。ncRNAの調節機構の一例を挙げると、ncRNAが標的のmRNAと結合することで、タンパク質の合成を抑制または活性化する現象が見つかっています(図1)。このような現象はRNA間相互作用と呼ばれていますが、相互作用の仕組みを解明するためには、2つのRNAから構成される結合構造を解析することが重要となります。
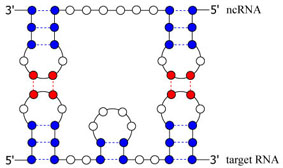
図1
RNA間相互作用の例。破線で結ばれた青丸の対は内部塩基対を表し、赤丸の対は外部塩基対を表す。
RNAの1本鎖は4種類の塩基A、C、G、Uからなる文字列(塩基配列)で表現され、その折り畳み構造は4種類の塩基が選択的に水素結合することで形成されます。この結合の仕組みは相互作用時における2つのRNA間の結合部位にも当てはまります。これまでいくつかの研究グループにより、RNA間相互作用予測問題に対して計算機に基づく様々なアプローチが提案されました。ここでは、2本のRNA塩基配列から結合構造や結合部位を予測することが目的となります。従来法では、複雑な結合構造を解析するときには計算効率が極端に悪くなるという欠点がありました。そのため、複雑な結合構造を扱うことができ、かつ高速に動作する予測手法が求められていました。
本研究では、高速かつ高精度のRNA間相互作用予測プログラムRactIP(ラクティップ)を開発しました。RactIPでは、結合構造の近似確率分布の下で予測構造の精度の期待値を最大化する問題を、閾値カットを用いた整数計画法で解きます(図2)。
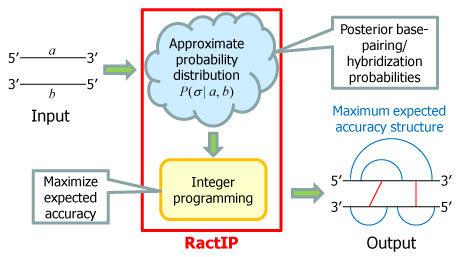
図2
RactIPの概観。
実際の相互作用データを用いた計算機実験結果では、RactIPは最新の既存手法と比べて同等以上の予測精度を達成しましたが、特筆すべきはその実行時間の劇的な短縮にあります。既存手法で結合構造予測に4000秒必要としたデータがRactIPでは0.1秒で済むなど、最大で約4万倍の高速化を実現しました。なお、RactIPのプログラム(ソースコードと実行ファイル)はhttp://www.ncrna.org/software/ractip/から自由に利用できます。
本研究を発表した論文はバイオインフォマティクスの主要な国際会議であるECCB2010に採録されましたが、ECCB2010の採録率が17%未満だったことを考えても、本手法が当該分野に与える影響は大きいものであると著者一同信じてやみません。
本研究は一部科学研究費補助金(若手研究(B))の助成を受けて行われました。
 京都大学 化学研究所
京都大学 化学研究所 国際共同利用・共同研究拠点
国際共同利用・共同研究拠点